Геномная селекция – молодое, но очень перспективное направление развития современного животноводства в вопросе разведения скота мясного направления продуктивности. Использование ДНК-маркеров для прогнозирования генетических качеств позволяет ускорить процесс генетического улучшения за счет повышения точности генетической оценки. В статье представлены результаты исследования, посвященного полногеномному поиску ассоциаций с QTL мясной продуктивности у скота казахской белоголовой и аулиекольской пород.
Ранние попытки изучения генетический контроль количественных признаков у животных были связаны с обнаружением хромосомных областей или локусов количественных признаков (QTL). Появление наличию чипов для крупного рогатого скота идентификация значимых SNP обеспечило более точное картирование регионов QTL. Все эти исследования привели к появлению множества кандидатов на QTL, для развития мясного скотоводства была создана обширная база данных крупного рогатого скота. Однако генетическая архитектура, включая причинные варианты ДНК, все еще остается в значительной степени неизвестной.
Полногеномный поиск ассоциаций с QTL
В статье представлено исследование, проведенное с целью проведения полногеномного поиска ассоциаций c QTL мясной продуктивности у скота казахской белоголовой и аулиекольской пород. Для проведения анализа проводился забор крови у животных двух видов. Подробнее ознакомиться с проведением исследования и его результатами можно в журнале «Главный зоотехник» № 7, 2022 ИД «Панорама».
Обработку контроля качества данных генотипа, влияющего на мясную продуктивность скота,
проводили с помощью Plink по следующим фильтрам:
- частота вызовов по всем SNP для отдельной выборки не ниже 90%;
- частота встречаемости каждого из исследованных SNP для всех генотипированных образцов не менее 90%;
- частота минорного аллеля для каждого из исследованных SNP не ниже 5%;
- отклонение генотипов SNP от распределения Харди – Вайнберга в наборе тестируемых образцов с p-значением 10-3.
Полногеномное ассоциативное исследование (GWAS) в мясном скотоводстве также было выполнено с использованием Plink. Форматирование полученных файлов и визуализация производились с помощью интерпретируемого языка программирования Python.
Показатели роста крупного рогатого скота
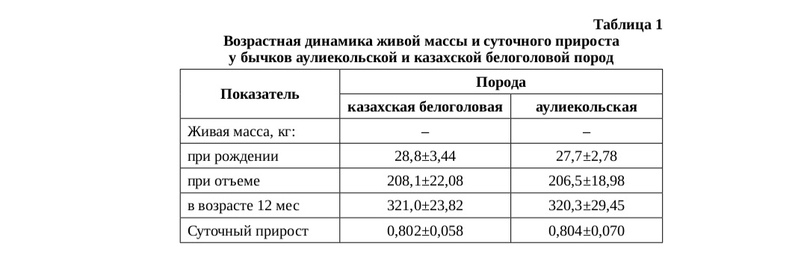
Для улучшения продуктивности мясного направления крупного рогатого скота был проведен GWAS-анализ четырех показателей роста бычков двух пород: казахской белоголовой и аулиекольской. В табл. 1 можно ознакомиться с показателями возрастной динамики живой массы и суточного прироста у бычков. Для аулиекольской породы были обнаружены значимые ассоциации для живой массы при рождении, при отъеме и живой массы животных в возрасте 12 мес. В свою очередь, у казахской белоголовой породы большинство значимых ассоциаций были связаны с признаком суточного прироста живой массы.
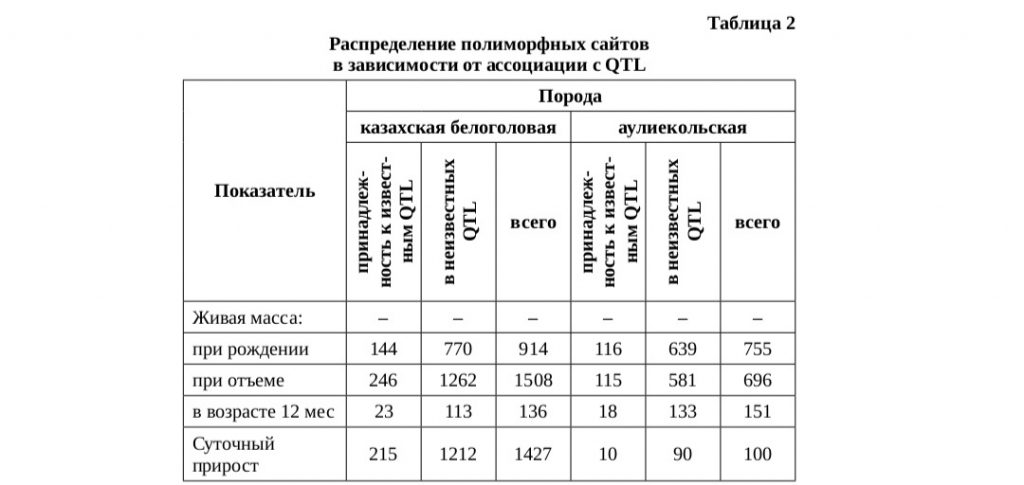
По данным табл. 2 можно отметить, что процент полиморфных сайтов в среднем для всех анализируемых параметров обеих пород составил 14,7. В результате установлено, что подавляющее большинство расположено вне известных и описанных QTL.
Разность между числом QTL-ассоциированных и неассоциированных полиморфных сайтов и неописанных на сегодняшний день говорит о необходимости их детального изучения с целью получения новых данных о роли выявленных сайтов в реализации фенотипических параметров, изучаемых в молочном и мясном скотоводстве.
Распределение полиморфных сайтов в хромосомах
Дальнейший этап исследования был посвящен получению более подробных данных о хромосомах, несущих не только наибольшее количество полиморфных сайтов, вошедших в модель, но и разделенных на QTL-ассоциированные и неассоциированные. Для ряда пород полиморфные сайты распределяются по всем хромосомам сравнительно равномерно. При этом распределению собственно QTL-ассоциированных сайтов уделяется недостаточное внимание.
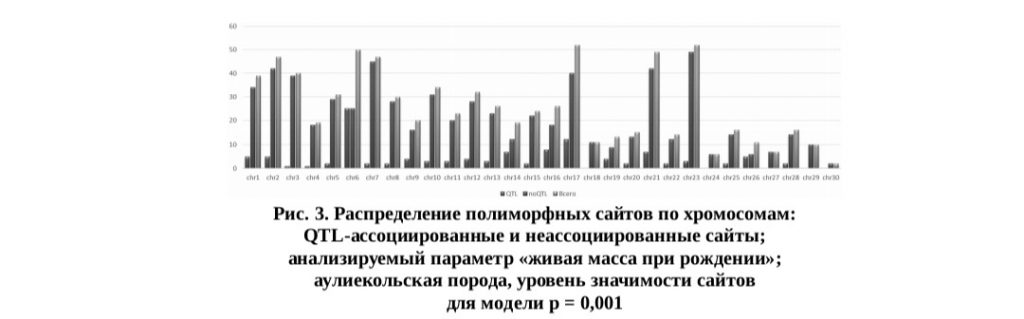
На рис. 3 представлено распределение полиморфных сайтов. По количеству QTL ассоциированных сайтов для параметра «живая масса при рождении» у бычков аулиекольской породы мясного скотоводства в России выделяется хромосома № 6, а далее по вкладу хромосома № 17. Также нужно обратить внимание на хромосомы № 14, 16 и 21. По количеству QTL неассоциированных сайтов лидирует хромосома № 23, а внимания заслуживают хромосомы № 2, 7, 17 и 21.
Сложно выделить какую-то особую хромосому или их группу, а отмеченные для аулиекольской породы хромосомы № 6 и № 17 в этом случае не выделяются. Напротив, хромосомы № 7 и № 21, сосредоточившие в себе максимально число QTL-неассоциированных сайтов у аулиекольской породы в случае казахской белоголовой характеризовались низким процентным содержанием этих сайтов.
Полученные данные говорят о том, что подавляющее большинство статистически значимых полиморфных сайтов у обеих пород расположено вне известных и описанных QTL. Разность между числом QTL-ассоциированных и неассоциированных полиморфных сайтов и неописанных на сегодняшний день говорит о необходимости их детального изучения с целью получения новых данных о роли выявленных сайтов в реализации изучаемых фенотипических параметров скотоводства мясного направления.
Особый интерес представляет факт, что, несмотря на то, что казахская белоголовая порода встречается во многих хозяйствах мясного скотоводства в большем числе районов и стран, степень описания QTL для параметра набора живой массы в данном случае еще ниже, чем у аулиекольской породы.
С помощью этого исследования был проведен полногеномный поиск ассоциаций c QTL скота мясного направления продуктивности казахской белоголовой и аулиекольской пород, что может помочь сократить интервалы генераций и значительно ускорить генетический прогресс в животноводстве.
Ознакомиться с полной версией материала и другими публикациями вы можете, оформив подписку на журнал «Главный зоотехник» ИД «ПАНОРАМА».